零基础入门转录组分析——数据处理(GEO数据库——高通量测序数据)
零基础入门转录组分析——数据处理(GEO数据库——高通量测序数据)
目录
- 零基础入门转录组分析——数据处理(GEO数据库——高通量测序数据)
- 1. 数据集获取
- 2. 数据处理(Rstudio)
- 3. 数据标准化(Rstudio)
- 注:配套资源只要改个路径就能运行,本人已检测过可以跑通,请放心食用,食用过程遇到问题,可先自行百度,实在解决不了可以私信
GEO数据库全称GENE EXPRESSION OMNIBUS,是由美国国立生物技术信息中心NCBI创建并维护的基因表达数据库。它创建于2000年,收录了世界各国研究机构提交的高通量基因表达数据,也就是说只要是目前已经发表的论文,论文中涉及到的基因表达检测的数据都可以通过这个数据库中找到。
并且GEO网站这个网站作为各种高通量实验数据的公共存储库。这些数据包括基于单通道和双通道微阵列的实验,检测mRNA,基因组DNA和蛋白质丰度,以及非阵列技术,如基因表达系列分析(SAGE),质谱蛋白质组学数据和高通量测序数据。可以按照文献数据集编号等众多形式进行检索。但是在这篇教程中仅介绍如何从GEO网站上根据数据集编号下载所需要的GEO数据集,并且下载后在R中对数据集进一步处理成后续分析所要的形式。
本项目以妊娠期糖尿病GSE154414数据集(高通量测序数据)作为展示 选用的数据库是GEO。 实验分组:疾病组,对照组。 我这里使用的R版本是4.2.2 要用到的R包:tidyverse,GEOquery
1. 数据集获取
首先进入GEO网站官网(如下图所示),在检索位置输入数据集编号,点击箭头指向的位置进一步运行搜索。
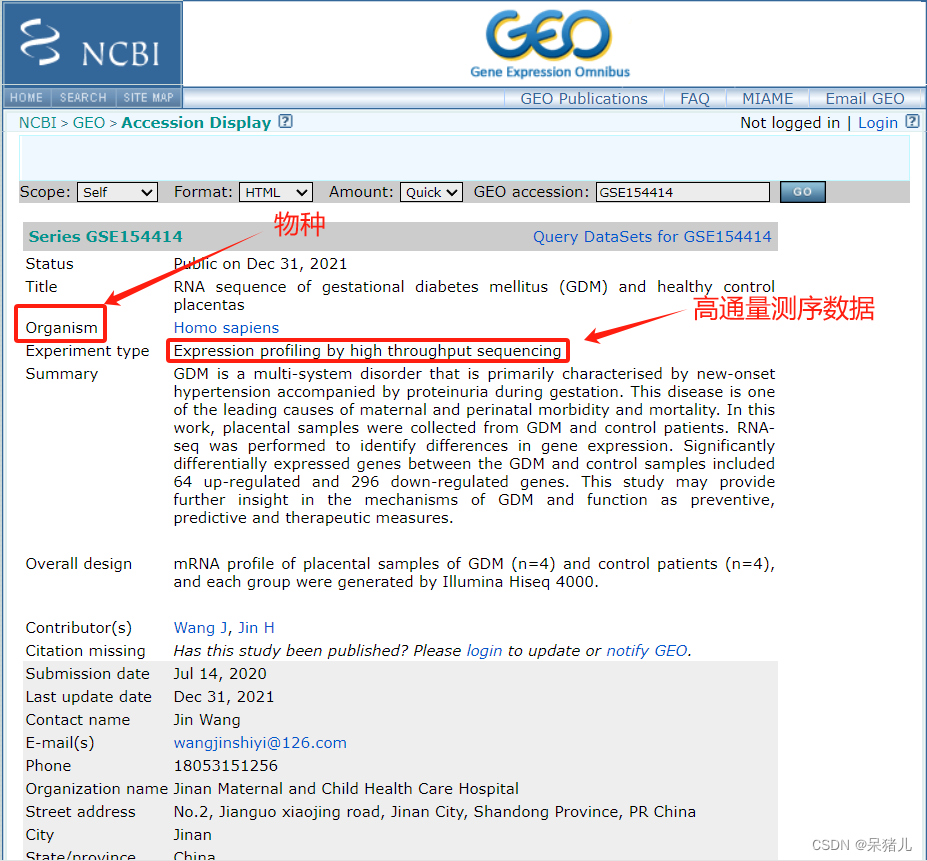
搜索之后会弹出如下界面:首先需要检查物种类型(Homo sapiens),之后查看数据集的类型是否是高通量测序/芯片数据,我这里是高通量测序数据(Expression profiling by high throughput sequencing),页面往下拉。
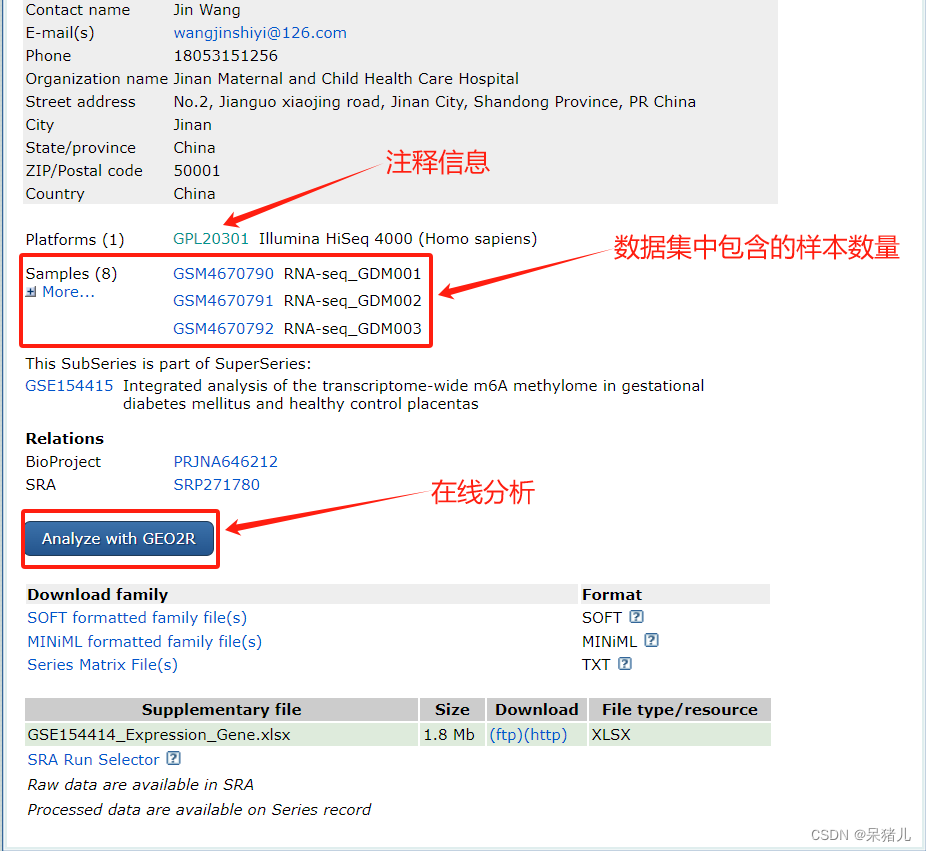
如下图所示:包含了该数据集对应的注释文件GPL20301,并且还列出来了数据集中包含的样本。
注:但是注释信息可以不用过多的关注,因为后续分析用不到,样本数量可以大致瞅一眼
到此对于该数据集已经有了初步了解(实际上就是看是不是高通量测序数据),如果是高通量测序数据就按照下面的操作进行,如果是芯片数据,可以参考之前的教程零基础入门转录组分析——数据处理(GEO数据库——芯片数据)
2. 数据处理(Rstudio)
rm(list = ls()) # 删除工作空间中所有的对象 setwd('/XX/XX/XX') # 设置工作路径 if(!dir.exists('./00_rawdata')){ dir.create('./00_rawdata') } # 判断该工作路径下是否存在名为00_rawdata的文件夹,如果不存在则创建,如果存在则pass setwd('./00_rawdata/') # 设置路径到刚才新建的00_rawdata下加载包:
library(GEOquery) library(tidyverse)
标注一下需要下载的数据集编号,并且在当前00_rawdata文件夹下创建一个名为GSE154414的文件夹(这是为了方便管理,如果不单独创建文件夹,数据集很多的话,就会显得很乱)
GEO_data dir.create(paste0(GEO_data)) } setwd(paste0(GEO_data)) N expr_fpkm[which(expr_fpkm print("log2 transform not needed") }